CITA ESTE TRABAJO
Muñoz-García-Borruel M, Álvarez-Barrientos A, Muñoz-Sanz A, Gutiérrez-Martín Y. Tetraspaninas y miRNAs en vesículas extracelulares urinarias en pacientes con pólipos colónicos e historia familiar de cáncer colorrectal. RAPD 2025;48(4):146-152. DOI: 10.37352/2025484.1
Listado de abreviaturas
AF CCR: antecedentes familiares de cáncer colorrectal.
CCR: cáncer colorrectal.
CF: citometría de flujo.
DE: desviación estándar.
miRNA: microRNA.
qPCR: PCR cuantitativa a tiempo real.
RIC: rango intercuartílico.
VE: vesículas extracelulares.
Introducción
El cáncer colorrectal (CCR) ocupa la tercera posición en incidencia y mortalidad de ambos sexos[1]. Gracias al diagnóstico precoz y a la implantación de programas de cribado poblacional, la mortalidad ha disminuido. Sin embargo, debido a los hábitos de vida y a la falta de recursos en los sistemas sanitarios, el CCR sigue siendo un importante problema de salud pública. Aunque la mayoría de los CCR son esporádicos, en torno al 15% presentan antecedentes familiares de CCR (AF CCR)[2]. Por lo tanto, en las personas con AF CCR de primer grado el diagnóstico precoz de lesiones preneoplásicas es mandatorio.
Una tendencia actual es la búsqueda de pruebas no invasivas dirigidas a diagnosticar pacientes con lesiones precursoras o a estratificar a los grupos de riesgo. Y, dentro de ellas, se incluyen los biomarcadores (biopsia líquida) que se pueden analizar en multitud de fluidos biológicos. La orina es una excelente muestra por su facilidad de recogida y almacenamiento y por su abundancia en metabolitos y otras moléculas. Un modo de aproximarse a este tipo de investigación es el estudio de las vesículas extracelulares (VE) y de las moléculas que transportan. Las VE son muy abundantes en el organismo y constituyen vehículos de información intercelular [3],[4].
Dentro del cargo de las VE destacan los microRNAs o miRNAs y unas proteínas de membrana, las tetraspaninas, que podrían ser marcadores de las VE, ya que además de formar parte de la estructura de la membrana, participan en la génesis, distribución, transporte y eliminación de los componentes del cargo[5]-[8].
Se han identificado multitud de miRNAs que parecen estar implicados en el desarrollo de pólipos y CCR[9]. Los miRNAs pueden tener actividad oncogénica (onco-miRNAs) o anti-oncogénica (anti oncomiRNAs)[10]-[12]. Entre los onco-miRNAS destacan: el miR-31 (estimulador de KRAS), el miR-21, con propiedades oncogénicas a través de la represión de la diana PDCD4 (Programmed Cell Death 4) (supresor tumoral proinflamatorio), y el miR-200, de importancia en el mantenimiento de la identidad epitelial que reprime factores de transcripción promesénquima y promueve la metástasis. Por otro lado, entre los miRNAs con funciones anti-oncogénicas destacan: la familia de miR-34, clave en la represión de la migración tumoral, la invasión y formación de metástasis y la familia miR let-7, incluida entre los más importantes en la represión de la oncogénesis debido a su abundancia y su función antiproliferativa.
Respecto a la biopsia líquida, la orina es un excelente fluido biológico que ofrece importantes ventajas para su análisis: es una muestra asequible y cómoda para el investigador y para el paciente, reproducible, poco estudiada y es abundante en VE y en miRNAs procedentes de diferentes orígenes del organismo, entre ellos, el colon.
Los objetivos de este trabajo fueron analizar el perfil de tetraspaninas por citometría de flujo (CF), establecer miRNAs urinarios que pudieran ser útiles como biomarcadores de patología colónica neoplásica o preneoplásica y demostrar el valor práctico de la orina en el análisis de las VE y del cargo intravesicular.
Pacientes y métodos
Se trata de un estudio observacional analítico prospectivo que incluyó a sujetos sometidos a colonoscopia por AF CCR de primer grado con edades entre 18-70 años y sujetos a los que se le realizó colonoscopia de cribado (grupo control). El estudio se llevó a cabo entre noviembre de 2019 y noviembre de 2020. A todos los pacientes se les extrajo una muestra de orina el día de la colonoscopia.
Los criterios de exclusión fueron: edad <18 años o >70 años y personas con hallazgos endoscópicos de colitis infecciosa/isquémica, enfermedad inflamatoria intestinal o CCR.
Las colonoscopias debían cumplir los criterios de calidad recomendados. Los pólipos resecados se remitieron al servicio de Anatomía Patológica de cada centro.
El día de la colonoscopia se extrajo una muestra de orina (10-20 ml) y se almacenó en un congelador a -80ºC hasta su análisis. Posteriormente, las muestras se remitieron al laboratorio del Servicio de Técnicas Aplicadas a la Biociencia de la Universidad de Extremadura para su análisis por CF y secuenciación masiva. Las muestras seleccionadas se identificaron mediante un código numérico.
Los datos de cada paciente se archivaron con las medidas de seguridad adecuadas en cumplimiento con la Ley Orgánica 15/1999 de Protección de Datos.
Análisis estadístico
Las variables cualitativas se presentaron mediante tablas de frecuencias y las cuantitativas se resumieron en su media y desviación estándar (DE) o su mediana y rango intercuartílico (RIC) para variables que no siguieron una distribución normal. Se utilizó IBM SPSS© v. 20.0.0.
Metodología en el laboratorio y análisis bioinformático
Se centrifugaron y filtraron las muestras urinarias para obtener las VE haciendo dos alícuotas: una para el estudio de tetraspaninas mediante análisis por CF y otra para la extracción de los miRNAs y su secuenciación masiva utilizando la tecnología de Ion Torrent.
Para el estudio de las tetraspaninas en las membranas de las VE se realizó una tinción con Carboxifluoresceína Succinimidyl Ester (CSFE) para analizar la calidad de la preparación y la otra muestra se usó para teñirla con los anticuerpos antiCD9, antiCD63 y antiCD81 conjugados con los diferentes fluorocromos y así visualizarlos por los canales correspondientes del citómetro de flujo (Cytoflex S, BeckmanCoulter).
Para la secuenciación de los miRNAs, primero se realizó la extracción de la población de los RNA de pequeño tamaño con el kit EasyPure miRNA (Transgenbiotech, China). Los miRNAs obtenidos se midieron con el Bioanalyzer de Agilent con el kit Bioanalyzer High Sensitivity RNA Analysis, (Agilent, USA). Tras comprobar que todos los parámetros de calidad eran buenos, se continuaba con la preparación de la librería para secuenciación masiva de la plataforma de Ion Torrent siguiendo el protocolo del Kit Qiaseq miRNA Library Kit (Qiagen, Alemania) con posterior templado en el IonChef (ThermoFisher, USA) con el kit Ion 550™ Kit—Chef (ThermoFisher, USA). Finalizada la carga del chip, se procedía a su lectura en el secuenciador Ion S5 XL con los reactivos específicos incluidos en el kit.
Tras la secuenciación de la librería se obtuvieron las secuencias de los miRNAs de cada muestra y se utilizaron para realizar una búsqueda en las bases de datos: miRBase v22genome-build-id: GRCh38, genome-build-accession: NCBI_Assembly: GCA_000001405.15), Bsgnenome.Hsapiens.UCSC.hg38.masked, https://bioconductor.org/packages/release/data/annotation/html/BSgenome.Hsapiens.UCSC.hg38.masked.html (genoma de referencia) de cara a identificar cuáles eran los miRNAs presentes en cada muestra y cuántas eran copias. Posteriormente se realizó un análisis bioinformático para comparar los miRNAs presentes en cada grupo para identificar aquellos miRNAs que aportasen información sobre la predicción de la aparición de lesiones colorrectales.
Una vez identificados los miRNAs de interés por su sobreexpresión o infraexpresión, se diseñaron sondas Taqman para cada uno de ellos compatibles para la química de ThermoFisher cDNA Taqman Advanced miRNA Chef (ThermoFisher, USA) que se usó posteriormente para comprobar si esa misma información se podría obtener por PCR cuantitativa en tiempo real (qPCR), ya que esta técnica es más rápida y económica. Por ello, a partir de la preparación de VE urinarias utilizadas para el estudio de secuenciación masiva, se realizó nueva extracción de miRNA con el mismo protocolo que el descrito para la secuenciación. Con el pool de miRNAs obtenido y usando las sondas TaqMan se realizó la qPCR en un termociclador QuantStudio 6 (ThermoFisher, USA) siguiendo el protocolo descrito para el kit.
Resultados
Inicialmente se incluyeron 46 sujetos en el estudio con la obtención de 46 muestras de orina, pero, debido a la situación pandémica por Covid-19, solo se pudieron analizar 18 muestras, consideradas suficientes en la investigación como prueba de concepto. Las características basales de los pacientes, sus hábitos de vida y sus antecedentes se muestran en la tabla 1. De los 46 sujetos, 31 (69,39%) presentaban AF CCR de primer grado, 20 de ellos (43,47%) no tenían pólipos o eran pólipos hiperplásicos (grupo 1), 11 (23,91%) presentaban adenomas colorrectales (grupo 2) y el resto, 15 sujetos (32,60%) que eran los controles, carecían de antecedentes de primer grado de CCR y no presentaron pólipos.
Tabla 1
Características basales, hábitos de vida, consumo de tóxicos y fármacos y antecedentes familiares de tumores de los sujetos del estudio.
Todas las colonoscopias fueron completas, cumpliendo los estándares de calidad. La preparación fue adecuada (Boston ≥ 6) en 43 pacientes (93,48%). Respecto a la histología de los adenomas, solo uno fue avanzado (displasia de alto grado).
Análisis de las tetraspaninas
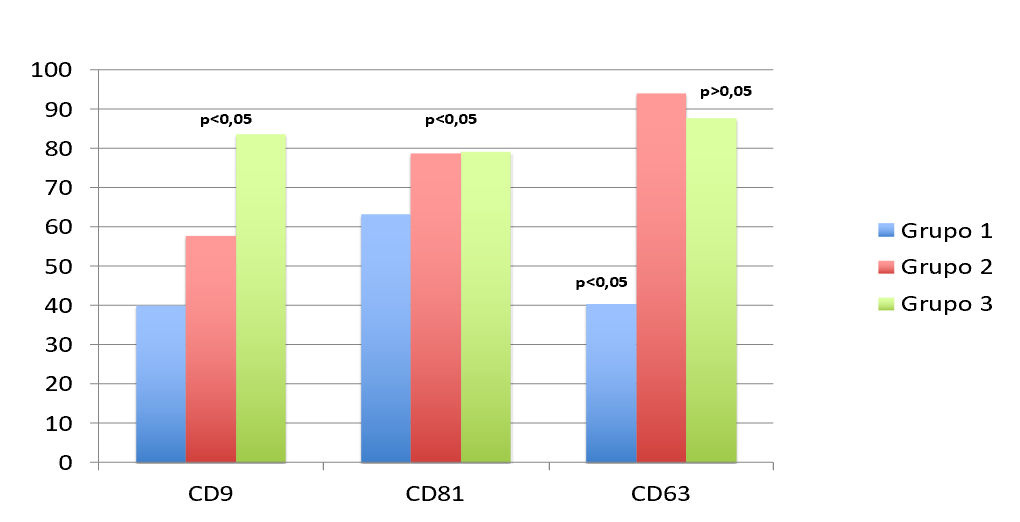
En la tabla 2 se muestran los valores descritos como porcentaje de positividad de las tetraspaninas CD9, CD81 y CD63 y en la figura 1 la comparación entre los tres grupos.
Tabla 2
Porcentaje de positividad de las tetraspaninas CD9, CD81 y CD63 expresados con sus medias±DE en cada grupo.
| Grupos | CD9 | CD81 | CD63 |
| Grupo 1 | 39,87±6,11 | 66,30±6,87 | 40,36±4,01 |
| Grupo 2 | 57,44±5,44 | 78,75±7,87 | 94,07±3,99 |
| Grupo 3 | 83,56±7,89 | 79,17±6,71 | 87,66±8,45 |
CD9: se observaron valores más bajos en ambos grupos con AF CCR respecto a los controles, con diferencias estadísticamente significativas (p<0,05).
CD81: se identificaron niveles más bajos en ambos grupos con AF CCR (grupos 1 y 2) comparados con los controles.
CD63: los niveles en el grupo 1 fueron más bajos que el de los controles, con diferencias estadísticamente significativas (p<0,05). En el grupo 2, los niveles fueron mayores que en los controles, aunque sin diferencias estadísticas.
Análisis de los microRNAs en las VE de orina
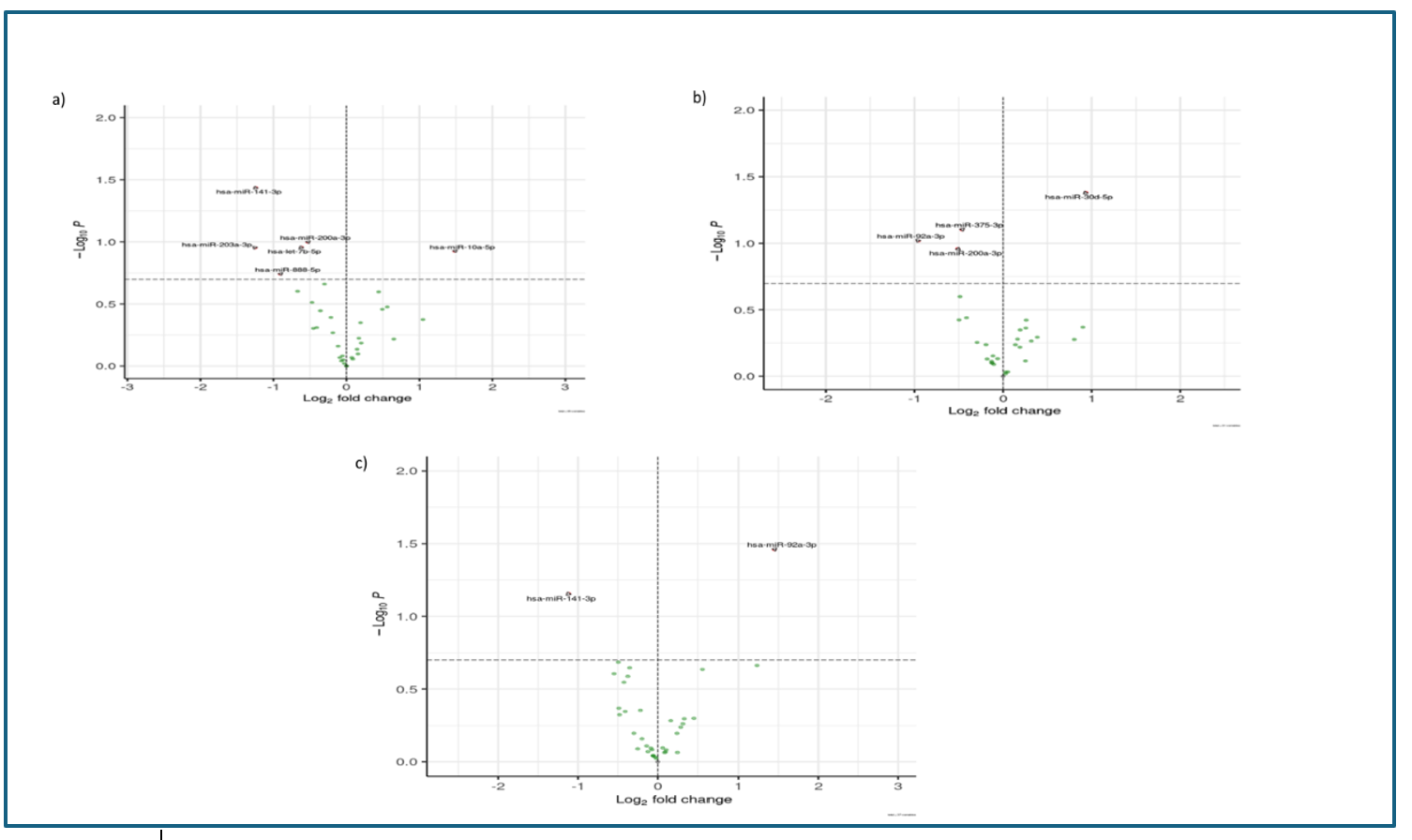
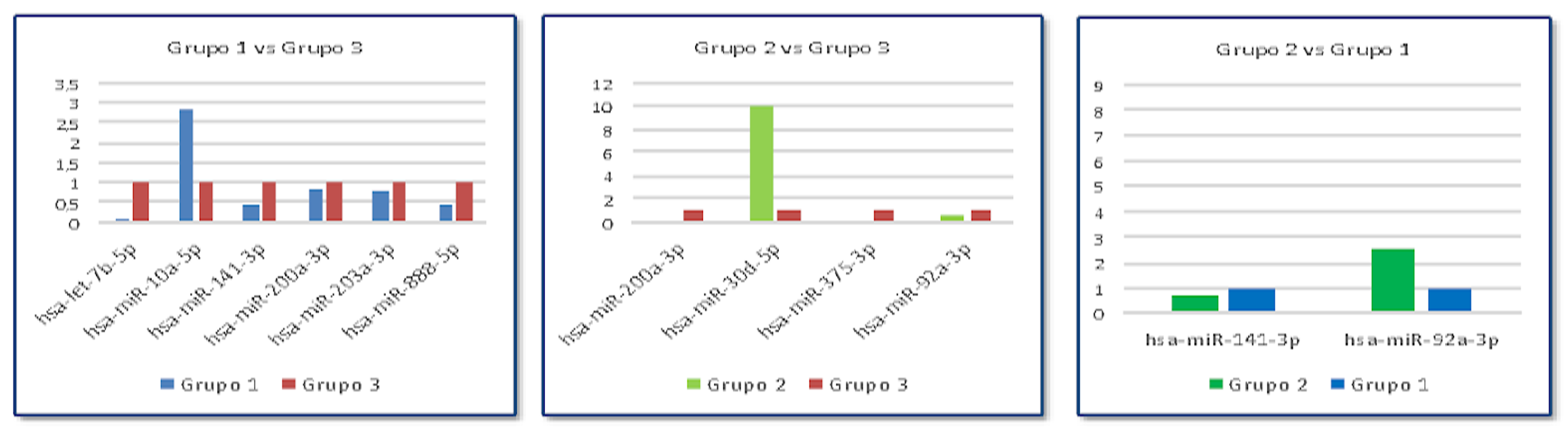
Para el análisis de los miRNAs presentes en las VE urinarias, tras el alineamiento de las secuencias obtenidas con las bases de datos de miRNAs publicados, se realizó la comparación entre grupos y se representaron en gráficos de volcán (Figura 2). Al comparar el grupo 1 y el grupo 3 se detectaron 6 miRNAs diferencialmente expresados. Destacan el hsa-mir-141-3p, expresado a la baja y con p<0.05, el miR-let-7b, que es un miRNA supresor oncogénico, también expresado a la baja y el hsa-miR-10a-5p que es un miRNA oncogénico y se encuentra sobreexpresado.
Figura 2
Gráficos de volcán. a) Comparación entre el grupo 1 y el grupo 3. b) Comparación entre el grupo 2 y el grupo 3. c) Comparación entre el grupo 2 y el grupo 1.
La comparación entre el grupo 2 y el 3 permitió detectar 4 miRNAs expresados diferencialmente. Sólo el hsa-miR-30d-5p presentaba una expresión elevada, estadísticamente significativa (p<0,05). El resto de miRNAs mostraron una expresión disminuida en el grupo de pacientes control, destacando el oncomiRNA hsa-miR-200a-3p.
Se hizo un estudio adicional comparando ambos grupos con AF CCR, encontrándose el hsa-miR-92-3p sobreexpresado en el grupo 2. Este mismo miRNA también se detectó sobreexpresado cuando se comparó con los controles.
De todos los miRNAs secuenciados, sólo 9 presentaron diferencias significativas entre los grupos (hsa-miR-92a-3p, hsa-miR-200a-3p, hsa-miR-141-3p, hsa-miR-203a-3p, hsa-miR-375-3p, hsa-miR-10a-5p, hsa-miR-30d-5p, hsa-miR-888-5p y hsa-let-7b-5). Éstos fueron los seleccionados para su estudio por qPCR usando sondas Taqman específicas para cada uno (Figura 3). Como genes normalizadores o "housekeeping" se utilizaron hsa-miR-186-5p y hsa-miR-191-5p, ya que se encontraban presentes en todas las muestras en la secuenciación masiva y sus niveles de expresión se mantenían por igual en todas ellas independiente del grupo. Se confirmaban las diferencias encontradas en la secuenciación masiva, es decir, en los casos en los que se identificaba una disminución o aumento de ese miRNA, se veía disminución o aumento en la expresión por qPCR.
Discusión
En la presente investigación analizamos los miRNAs vehiculados en las VE de orina, cuya función es regular la transcripción genómica. Un aspecto que conviene destacar es el papel de algunos miRNAs en la patogenia de los adenomas y del CCR. Es cada vez más abundante la literatura sobre los miRNAs detectados en diversos tipos de muestras biológicas. Por ejemplo, en la sangre, los miR-21, miR-92a y miR-17-3p[13]; en los tejidos, un incremento de 6 miRNAs de tejido normal a CCR: miR-18a, miR-18b, miR-431, miR-503, miR-1246 y miR-4417 y un descenso de otros 5 miRNAs: miR-133a, miR-375, miR-378, miR-422 y miR-479[14] con 3 miRNAs: miR-21, miR-29a y miR-135b sobreexpresados en los adenomas frente al tejido colónico normal[15]; y también en las heces (miR-21 y miR-106a, miR-92a y miR-106a)[16]. Respecto a las heces, que ofrecen un perfil de miRNAs muy parecido al del tejido colónico, Ahmed et al.[17] propusieron un panel de 12 miRNAs (miR-7, miR-17, miR-20a, miR-21, miR-92a, miR-96, miR-106a, miR-134, miR-183, miR-196a, miR-199a-3p, miR-214) con expresión elevada en el CCR, sobre todo en el CCR metastásico, comparado con los adenomas. Los mismos autores describieron 8 miRNAs (miR-9, miR-29b, miR-127-5p, miR-138, miR-143, miR-146a, miR-222, miR-938) que muestran una expresión menor en los pacientes con CCR.
El perfil de los miRNAs detectado en nuestro estudio no permite asegurar ninguna firma individual o colectiva (clúster) definitoria de lesión colorrectal, aunque al menos dos miRNAs (miR-141-3p y miR-30d-5p) se expresan diferencialmente en las VE de los familiares de primer grado de los pacientes con CCR comparados con el grupo control, existiendo diferencias entre ambos grupos patológicos. Además, el miR-92a-3p se encuentra sobreexpresado en pacientes con adenomas, mientras que el miR-141-3p se expresa a la baja (se asocia a 81 enfermedades en humanos entre las cuales se encuentra, en segundo lugar, el CCR)[18]. Este dato está en línea con diversos trabajos internacionales que definen perfiles de miRNAs diagnósticos, en especial de CCR, aunque, no se refieren a lesiones con potencial maligno ni a familiares de sujetos con CCR[19]-[22]. Aunque nuestro trabajo no permite extraer conclusiones generalizables, el bajo número de muestras es un hecho bastante común en la literatura, por lo que un estudio con mayor número de muestras permitiría definir miRNAs con expresión aumentada en las lesiones colorrectales pre o neoplásicas.
El perfil de expresión de las tetraspaninas (CD9, CD63 y CD81) detectadas por CF, que permite una detección rápida y eficiente, en los familiares de primer grado de los pacientes con CCR es diferente al del grupo control. En nuestra investigación detectamos un nivel menor de CD9 en los sujetos con AF CCR respecto a los controles. La tetraspanina CD9 está implicada en la adhesión celular y en la captación de VE a células tumorales colorrectales[23]. Estos procesos están negativamente regulados por la expresión de la tetraspanina CD9 en las VE. Las tetraspaninas CD81 y CD63 están involucradas en la progresión del CCR y, por otra parte, parece que la expresión de ambos se correlaciona con la invasión tumoral y las metástasis. En nuestro estudio, ambos grupos con AF CCR presentaron valores más bajos de CD81 comparados con los controles, los sujetos con AF CCR y adenomas presentaron niveles de CD63 mayores que en los controles y el grupo con AF CCR y sin adenomas mostró niveles inferiores. Un estudio con un mayor número de muestras tal vez podría definir un perfil diferencial propio de tetraspaninas en las VE urinarias para los distintos estadios de patología colorrectal.
Conclusiones
La presente investigación permite resaltar varios aspectos: 1) La validez de la orina como método diagnóstico adecuado para el estudio de las VE y de los miRNAs vehiculados en ellas. 2) El análisis de las tetraspaninas, que, aunque son proteínas utilizadas en la CF en calidad de marcadores de membrana, podrían complementar o sustituir a la determinación de los miRNAs; es decir, podría ser suficiente analizar el perfil de tetraspaninas en lugar de los miRNAs, lo cual requiere más tiempo, personal, recursos materiales y económicos al implicar tecnología genómica en lugar de la CF (más versátil, barata y rápida). Y 3) Esta prueba de concepto puede abrir una puerta para el posible diseño de un panel de sondas Taqman que, mediante qPCR, estudie la expresión de los miRNAs en cada sujeto de riesgo en busca de un diagnóstico preventivo no invasivo de lesiones colorrectales.

 Descargar número completo
Descargar número completo Download full issue
Download full issue